Стохастика разрушения: аутофаголизосомная система клетки
- Авторы: Кудряева А.1, Соколов А.1, Белогуров А.1,2
-
Учреждения:
- Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН
- Московский государственный университет им. М.В. Ломоносова
- Выпуск: Том 12, № 1 (2020)
- Страницы: 18-32
- Раздел: Обзоры
- Дата подачи: 30.03.2020
- Дата принятия к публикации: 30.03.2020
- Дата публикации: 16.04.2020
- URL: https://actanaturae.ru/2075-8251/article/view/10936
- DOI: https://doi.org/10.32607/actanaturae.10936
- ID: 10936
Цитировать
Аннотация
Аутофагия – консервативный эволюционно древний процесс, который обуславливает перемещение завершивших свою функцию, избыточных или потенциально опасных клеточных компонентов в лизосому для их последующей деградации. Этот процесс имеет важнейшее значение в рециркуляции энергии и субстратов, необходимых для клеточных процессов. Аутофагия играет одну из главных ролей не только в выживании клетки при стрессе, но также активно участвует в поддержании клеточного гомеостаза, влияет на иммунитет и занимает значительное место в клеточном ремоделировании в процессе развития организма. Эффективность аутофагии обеспечивается управляемым взаимодействием двух органелл – аутофагосомы и лизосомы. Несмотря на значительные успехи в описании молекулярных механизмов, опосредующих функционирование аутофаголизосомной системы (АЛС), достигнутые за последние два десятилетия ее активного изучения, множество фундаментальных вопросов все еще остаются открытыми: существуют ли лизосомы со специализированными функциями, какова роль АЛС в патогенезе заболеваний человека, таких, как нарушение метаболизма липидов, инфекции и старение. Понимание механизмов всех этапов аутофаголизосомной деградации – от инициации аутофагии до терминального этапа разрушения субстратов в лизосоме – позволит выработать новые подходы к направленному воздействию на АЛС и, как следствие, к контролю клеточного протеостаза.
Ключевые слова
Полный текст
СПИСОК СОКРАЩЕНИЙ
АЛС – аутофаголизосомная система; УПС – убиквитин-протеасомная система; ЭПР – эндоплазматический ретикулум.
ВВЕДЕНИЕ
Деградация белков – одна из основных внутриклеточных функций, которая регулирует множество важных процессов, тем самым обеспечивая клеточный гомеостаз и выживание всего организма в целом. Аутофаголизосомная (АЛС) и убиквитин-протеасомная системы (УПС) являются основными путями внутриклеточного протеолиза, уменьшение или увеличение эффективности функционирования которых существенно влияет на клеточный метаболизм в норме и при патологических процессах [1].
Контролируемый протеолиз короткоживущих, а также неправильно свернутых внутриклеточных белков осуществляется в основном УПС. Эта система опирается на координированные действия трех типов близкородственных ферментов: E1-, E2- и E3-лигаз, которые конъюгируют небольшой белок убиквитин (ubiquitin, Ub) с полипептидными субстратами, которые должны подвергаться деградации [2] (рис. 1). Мультисубъединичный протеолитический комплекс, называемый 26S протеасома, распознает белок, модифицированный подобным образом. После связывания субстрата убиквитиновая цепь высвобождается благодаря ассоциированным с протеасомой деубиквитинирующим ферментам (deubiquitinating enzyme, DUB), далее субстрат подвергается разворачиванию и затем транслоцируется во внутреннюю полость протеасомы, где расщепляется на короткие пептиды, которые могут экспонироваться на поверхности клеток либо дополнительно измельчаться до свободных аминокислот различными аминопептидазами [3]. В последнее время появляется все больше данных о возможности убиквитин-независимого гидролиза белков [4]. Первым белком, для которого показан подобный механизм деградации, была орнитиндекарбоксилаза [5]. Недавно на примере основного белка миелина – одного из основных аутоантигенов при рассеянном склерозе – обнаружен новый механизм опосредованного зарядом убиквитин-независимого гидролиза белков протеасомой [6, 7].
Рис. 1. Убиквитин-протеасомная система. Убиквитин синтезируется в виде четырех белков-предшественников – UBC, UBB, UBA52 и UBA80, которые далее подвергаются процессингу специализированными деубиквитинирующими ферментами – убиквитин-изопептидазами (DUB). Система убиквитинирования, включающая три типа убиквитин-лигаз (Е1 (два представителя), Е2 (десятки представителей) и Е3 (сотни представителей)), высокоспецифична и избирательна за счет построения по принципу иерархического усложнения. Убиквитин конъюгируется с субстратами (S) в виде мономера или полиубиквитиновой цепи, которая формируется через внутренние остатки лизина предшествующего Ub. Элонгацию полиубиквитиновой цепи обеспечивают лигазы Е3 или относительно недавно открытые убиквитин-лигазы семейства Е4. Между убиквитинированием и процессом удаления остатков убиквитина убиквитин-изопептидазами существует динамическое равновесие, которое обуславливает оптимальную длину цепи, по современным представлениям составляющую примерно шесть молекул убиквитина на молекулу субстрата [8]. Далее убиквитинированный субстрат связывается с субъединицами протеасомы Rpn10, Rpn13 и Rpn1 либо напрямую, либо с участием шаттл-белков семейства UBL–UBA. Определенное количество убиквитина попадает в протеолитическую камеру вместе с субстратом, что приводит к его разрушению. В подавляющем большинстве случаев резидентная протеасомная деубиквитиназа Rpn11 успешно удаляет всю полиубиквитиновую цепь, которая далее подвергается расщеплению на мономеры для повторного использования
Основное отличие АЛС от УПС состоит в том, что АЛС участвует в утилизации больших и потенциально опасных клеточных структур, например белковых агрегатов и органелл. В большинстве случаев АЛС-опосредованный протеолитический процесс (называемый также аутофагия) активируется в ответ на недостаток питательных веществ в клетке, и значительную роль в нем играют белки семейства ATG (Autophagy-related proteins) [9]. Наиболее изученным аутофагосомным процессом является макроаутофагия, при которой клеточные компоненты, предназначенные для деградации, захватываются аутофагосомами. Аутофагосомы представляют собой бислойные мембранные везикулы, которые образуются из предшественников, называемых фагофорами – областей цитоплазмы, изолированных мембраной, которые формируются и расширяются благодаря координированным действиям белков семейства ATG [10]. В дальнейшем аутофагосомы сливаются либо непосредственно с лизосомами, где происходит гидролиз их содержимого протеолитическими ферментами, либо сначала сливаются с эндосомами, формируя промежуточный компартмент, называемый амфизомой. Внутри лизосом цитоплазматический материал разлагается на метаболиты, которые повторно могут использоваться клеткой в качестве строительных блоков для синтеза новых макромолекул или источника энергии. Таким образом, аутофагия имеет решающее значение для метаболизма клетки, особенно в условиях голодания. Кроме того, удаление поврежденных или избыточных органелл, белковых агрегатов и патогенов способствует более продолжительной жизнедеятельности клетки [11]. Первоначально считалось, что процесс аутофагии является неселективным. Однако позже стало понятно, что сигналом деградации, как и в УПС, может служить модификация субстратов убиквитином [12].
Нарушения в функционировании УПС или АЛС могут быть как основной причиной, так и результатом многих патологических процессов. Старение, нейродегенеративные заболевания, такие, как болезни Альцгеймера (Alzheimer’s disease, AD), Паркинсона (Parkinson’s disease, PD) и Хантингтона (Huntington’s disease, HD), сердечно-сосудистые заболевания (включая атеросклероз), онкологические заболевания, заболевания иммунной системы (в том числе ревматоидный артрит) и мышечная дистрофия напрямую связаны с дефектами внутриклеточного протеолиза [13]. В этой связи знание молекулярной машинерии АЛС и путей ее регуляции приобретает особенно важное значение.
МЕХАНИЗМЫ И ТИПЫ АУТОФАГИИ
Аутофагия – эволюционно древний катаболический процесс, механизм которого консервативен у всех эукариотических клеток – от дрожжей [14] до млекопитающих [15]. Базальная (нестимулированная) аутофагия со стабильно низкой скоростью происходит во всех клетках, но может активироваться в тех случаях, когда клеткам необходимы питательные вещества и энергия (например, при голодании), при ремоделировании существующих или элиминации вредоносных компонентов цитоплазмы (например, при окислительном стрессе, инфекции или накоплении белков, вызванном стрессом ЭПР). Аутофагия опосредует деградацию окисленных липидов, поврежденных органелл (например, митохондрий и пероксисом), а также внутриклеточных патогенов (бактерий и вирусов). Посредством аутофагии разрушаются агрессивные агрегаты цитоплазматических белков при нейродегенеративных заболеваниях, таких, как различные формы деменции (вызванные белком тау), болезнь Паркинсона (α-синуклеин) и болезнь Хантингтона (мутантный хантингтин). Аутофагия защищает от некоторых инфекционных заболеваний, вызванных, например, Salmonella typhimurium и Mycobacterium tuberculosis. В результате деградации депонированного материала образуются нуклеотиды, аминокислоты и свободные жирные кислоты, из которых синтезируются макромолекулы и ATP. Наконец, аутофагия защищает клетки от возрастных изменений. Таким образом, этот сложный процесс, регулируемый многими факторами, участвует в защите клеток от злокачественной трансформации, инфекционных заболеваний, а также метаболических, мышечных, воспалительных и нейродегенеративных расстройств.
Как уже упоминалось во «Введении», изначально аутофагия считалась неселективным процессом деградации. Однако вскоре стало очевидно, что аутофагия может быть очень избирательной. Несмотря на растущий список субстратов, которые выборочно обрабатываются аутофагосомами, точные механизмы распознавания субстрата при аутофагии все еще недостаточно изучены. При дефиците питательных веществ или факторов роста процесс аутофагии является неселективным. Селективная и неселективная аутофагии инициируются различными сигналами. Тем не менее, все они инициируют ремоделирование мембраны, необходимое для образования аутофагоcомы.
На настоящий момент идентифицированы три типа аутофагии: микроаутофагия; аутофагия, опосредованная шаперонами (CMA, chaperone-mediated autophagy), которая встречается только у млекопитающих; и макроаутофагия.
Микроаутофагия – наименее изученный вид аутофагии (рис. 2). Этот вид аутофагии предложено подразделять на три типа: микроаутофагия с лизосомным выпячиванием (тип I), микроаутофагия с лизосомной инвагинацией (тип II) и микроаутофагия с эндосомной инвагинацией (тип III) [16]. В микроаутофагии типа I участвуют белки ATG5 (у растений), Vac8 и ATG18 (у дрожжей Pichia pastoris). Микроаутофагия типа III обнаружена сравнительно недавно и изучалась на линии дендритных клеток мыши и на Drosophila melanogaster. В микроаутофагии этого типа участвуют некоторые белки эндосомного комплекса сортировки (endosomal sorting complexes required for transport, ESCRT), такие, как Nbr1 и HSC70. В целом, микроаутофагия способствует прямой доставке в лизосомы органелл и других клеточных компонентов, например, пероксисом (микропексофагия), ядерных компонентов (фрагментарная микроаутофагия ядер) и митохондрий (микромитофагия). Аутофагия данного типа может активироваться не только в условиях голодания, но и в нормальных условиях, при этом деградации подвергаются неповрежденные компоненты клетки.
Рис. 2. Виды аутофагии. Наименее изученный вид аутофагии – микроаутофагия – способствует прямой доставке органелл и других клеточных компонентов в лизосомы. При шаперон-опосредованной аутофагии распознавание грузов шапероном HSPA8/HSC70 происходит благодаря наличию в их последовательности сигнального пентапептида KFERQ. Шаперон с грузом связывается с лизосомным мембранным белком 2A (LAMP2A), после чего груз транслоцируется в лизосомную полость. При макроаутофагии (или просто аутофагии) ATG-белки привлекаются к месту сборки фагофора (PAS), где происходит формирование изолирующей мембраны, которая и образует фагофор. Удлинение изогнутой изолирующей мембраны и дальнейшее ее закрытие приводят к образованию двойных мембранных везикул – аутофагосом, которые захватывают клеточный материал. Затем аутофагосома сливается с лизосомной мембраной, образуя аутолизосому. Это слияние приводит к деградации аутофагосомы вместе с клеточным материалом в лизосомной полости
При шаперон-опосредованной аутофагии цитозольные белки, содержащие определенную сигнальную последовательность – пентапептид KFERQ, распознаются белком теплового шока 70 кДа (HSPA8/HSC70), который, в свою очередь, связывается с лизосомным мембранным белком 2A (LAMP2A). Затем белки-мишени подвергаются разворачиванию и далее транслоцируются в лизосомный люмен, где и разрушаются [17] (рис. 2).
При индукции макроаутофагии (далее называемой просто аутофагией) происходит привлечение ATG-белков к месту сборки фагофора (phagophore assembly site, PAS), который представляет собой изолирующую мембрану чашеобразной формы. Постепенное удлинение изогнутой изолирующей мембраны приводит к расширению фагофора. В конечном итоге мембрана закрывается с образованием двойных мембранных везикул – аутофагосом. Размеры аутофагосом варьируют в пределах 0.5–1.5 мкм в зависимости от сигнала, индуцирующего аутофагию, груза (cargo), который необходимо подвергнуть деградации, и типа клетки [11]. После доставки по микротрубочкам к лизосоме мембрана аутофагосомы сливается с лизосомной мембраной, образуя аутолизосому. Это слияние приводит к деградации аутофагосомы вместе с грузом в лизосомной полости (рис. 2).
СТАДИИ АУТОФАГИИ
Процесс аутофагии состоит из нескольких стадий: инициация, образование аутофагосомы, расширение и удлинение мембраны аутофагосомы, замыкание мембраны, слияние аутофагосомы с лизосомой и деградация содержимого (рис. 3) [18].
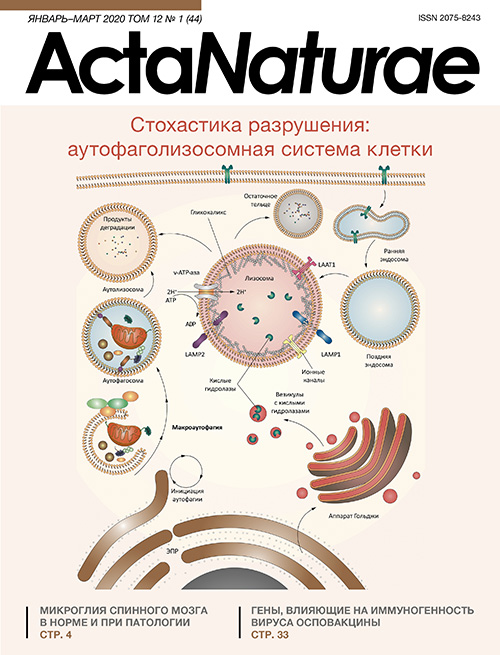
Рис. 3. Аутофаголизосомная система. Инициация аутофагии происходит при различных стрессовых условиях, таких, как голодание, гипоксия, окислительный стресс, агрегация белка, стресс эндоплазматического ретикулума и т.д. Основной инициаторный комплекс ULK1, состоящий из белков ULK1, ATG13, FIP200 и ATG101, инициирует нуклеацию фагофора с помощью фосфатидилинозитол-3-киназного комплекса I (PI3KC3–C1) класса III, в состав которого входят ATG14, Beclin-1, Vps34 и AMBRA1 и фактор везикулярного транспорта p115, который активирует продукцию фосфатидилинозитол-3-фосфата (PI3P) в омегасоме – субдомене мембраны эндоплазматического ретикулума. Затем PI3P привлекает белки WIPI2 и DFCP1 в омегасому через их взаимодействие с PI3P. Недавно было показано, что WIPI2 напрямую связывает ATG16L1, рекрутируя комплекс ATG12– ATG5–ATG16L1, который усиливает опосредованную ATG3 конъюгацию белков семейства ATG8, включая белки LC3 и GABARAP, с фосфатидилэтаноламином (PE), образуя таким образом связанные с мембраной липидные формы. ATG8 не только дополнительно привлекает компоненты аутофагосомной машинерии, содержащие LC3-взаимодействующую область (LIR), он необходим также для наращивания и закрытия мембраны фагофора. При селективной аутофагии LC3 участвует в секвестрации маркированного груза в аутофагосомы благодаря LIR-содержащим грузовым рецепторам. Некоторые клеточные мембраны, включая плазматическую мембрану, митохондрии, эндосомы и комплекс Гольджи, способствуют наращиванию аутофагосомной мембраны, передавая собственный мембранный материал (часть этих липидных бислоев поставляется ATG9-содержащими везикулами, но происхождение остальной части липидного бислоя в настоящее время неизвестно). Закрытие аутофагосомной мембраны приводит к образованию двухслойной везикулы, называемой аутофагосомой, которая созревает (происходит удаление белков ATG) и, наконец, сливается с лизосомой. Лизосомные кислые гидролазы подвергают деградации аутофагический груз, а затем питательные вещества высвобождаются в цитоплазму для повторного использования
Инициация
Стадия инициации регулируется различными белками в зависимости от исходного сигнала, индуцирующего аутофагию. К ним относятся четыре протеинкиназы – mTORC1, ULK1, AMPK и AKT. Голодание является одним из наиболее изученных факторов индукции аутофагии, при этом серин/треониновая киназа mTOR, входящая в состав комплекса mTORC1, играет значительную роль в определении доступности питательных веществ. Недостаток питательных веществ, в основном аминокислот, запускает сигнальный каскад, который ингибирует активность mTORC1 [19]. Неактивный mTORC1 диссоциирует от ULK1, что приводит к дефосфорилированию и активации комплекса ULK1 (также известного как ATG1), включающего в себя белки ULK1, ULK2, ATG13, FIP200 (RB1CC1) и ATG101. Комплекс ULK1 запускает формирование фагофора с помощью фосфорилирования компонентов фосфатидилинозитол-3-киназного комплекса I (PI3KC3–C1) класса III, в состав которого входят белки VPS34/PIK3C3, ATG14L, AMBRA1, Beclin-1 – ортолог ATG6, и транспортный фактор p115, который активирует образование фосфатидилинозитол-3-фосфата (PI3P) в специфической структуре эндоплазматического ретикулума, называемой омегасомой.
Рост мембраны
После образования комплекса ULK1 фосфатидилинозитол-3-киназный комплекс I (PI3KC3–C1) класса III рекрутируется к фагофору. Комплекс PI3K необходим для нуклеации и сборки изолирующей мембраны. Его основной компонент белок VPS34 – каталитическая субъединица комплекса PI3K, рекрутируется ULK1 и продуцирует PI3P в местах инициации. PI3P критически важен для формирования аутофагосом и считается маркером аутофагосомных мембран.
Фосфатидилинозитол-3-фосфат, продуцируемый на месте формирования фагофора, создает платформу для привлечения нижестоящих аутофагосомных эффекторов, таких, как белки семейства WIPI и ATG16L. Белок WIPI2 непосредственно связывает ATG16L1, привлекая комплекс ATG12–ATG5–ATG16L1, который способствует конъюгации семейства убиквитин-подобных (ubiquitin-like, UBL) белков ATG8, к которым относятся белки LC3 и GABARAP, с расположенным на мембране фагофора фосфатидилэтаноламином (PE), образуя мембраносвязанные липидированные формы белка. Модифицированные таким образом белки ATG8 в свою очередь дополнительно привлекают компоненты, которые содержат LC3-взаимодействующую область (LIR, LC3-interacting region), что способствует удлинению и закрытию мембраны фагофора. Конъюгированная с липидами форма белка LC3 может служить маркером аутофагосом. Помимо прочего, при селективной аутофагии LC3 участвует в доставке специфически меченного груза в аутофагосомы через LIR-содержащие грузовые рецепторы.
Важно отметить, что аппарат Гольджи, плазматическая мембрана и эндосомы также могут участвовать в аутофагосомном биогенезе [11], способствуя удлинению аутофагосомной мембраны путем донорства мембранного материала (часть липидов доставляется ATG9-содержащими везикулами, но происхождение остального липидного бислоя в настоящее время неизвестно).
Распознавание аутофагосомного груза
Селективная аутофагия имеет фундаментальное значение для метаболизма клетки. При селективной аутофагии рецепторы распознают груз (cargo) и присоединяют его к зарождающейся аутофагосоме (рис. 4). Рецепторы имеют в своем составе LC3-взаимодействующую область, которая содержит консенсусную последовательность Trp/Phe/Tyr-x-x-Leu/Ile/Val (W/F/YxxL/I/V), связывающуюся с семейством UBL-белков LC3/GABARAP, экспонированных на аутофагосомной мембране [20, 21]. Новые данные свидетельствуют о том, что связанные с грузом аутофагосомные рецепторы могут локально инициировать аутофагию путем рекрутирования и активации важнейших компонентов аутофагосомной системы (например, комплекса ULK1) [22, 23]. Это отличается от аутофагии, вызванной нехваткой питательных веществ, где инициация аутофагии и формирование аутофагосомной мембраны не зависят от груза, но регулируются протеинкиназами [11].
В зависимости от типа поглощаемого клеточного материала селективную аутофагию подразделяют на агрефагию (агрегированные белки), митофагию (митохондрии), пексофагию (пероксисомы), липофагию (липидные капли), рибофагию (рибосомы), ретикулофагию (ЭПР), ксенофагию (патогены), гликофагию (гликоген), зимофагию (зимоген), нуклеофагию (ядро), хроматофагию (хроматин), миелинофагию (миелин), ферритинофагию (ферритин), лизофагию (лизосомы), гранулофагию (стресс-гранулы) и протеафагию (протеасома) [16, 17].
В дополнение к связыванию с аутофагосомными мембранами рецепторы должны распознавать груз, т.е. отличать нормальные органеллы или клеточные структуры от поврежденных или избыточных [24]. У высших эукариот связывание может обуславливаться убиквитинированием груза. Этот механизм фактически является основной формой распознавания грузов у млекопитающих [25]. Помимо Ub-зависимого пути доставки в аутофагосому существует также и Ub-независимый. Часто бывает, что доставка одного и того же груза происходит с использованием обоих механизмов [20, 25, 26].
Рис. 4. Аутофагосомные рецепторы. Процессы селективной аутофагии называют в зависимости от типа поглощаемого клеточного материала. Рецепторы некоторых аутофагосомных грузов на настоящий момент не известны. Отдельные аутофагосомные рецепторы участвуют в доставке нескольких грузов, например p62 и NBR1
Убиквитин-зависимая аутофагия
Известно, что убиквитинированные белки накапливаются при ингибировании УПС и образуют агрегаты, которые утилизируются посредством аутофагии [20]. На настоящий момент описано около 20 разновидностей селективной аутофагии [19, 23] и почти половина из них – Ub-зависимые. При Ub-зависимой аутофагии клеточные компоненты для их доставки к аутофагосоме подвергаются модификации Ub, который в свою очередь распознается рецептором, содержащим UBD (убиквитинсвязывающий домен) [21, 27]. В клетке имеется большое количество аутофагосомных рецепторов для распознавания внутриклеточных убиквитинированных агрегатов (p62, NBR1, OPTN, TOLLIP) [28–32], бактерий (p62, OPTN, NDP52) [33–35], пероксисом (NBR1) [36], митохондрий (OPTN, NDP52, Tax1BP1) [23, 37, 38], зимогенов (p62) [39], протеасом (RPN10) [40], экваториальных пластинок (midbody) (p62, NBR1) [41] или нуклеиновых кислот (p62, NDP52) [42, 43] и связывания груза с аутофагосомными мембранами (рис. 4). Существует предположение, что способность убиквитинированных белков образовывать агрегаты, превращаясь тем самым в аутофагосомные субстраты, зависит от длины и типа Ub-цепи [44]. Имеются экспериментальные данные о повышенном сродстве полиубиквитиновых цепей K63 к аутофагосомным рецепторам p62 и NBR1 [45, 46], тогда как белки, модифицированные цепями K48, K27 и К11, подвергаются гидролизу протеасомой [47].
Агрефагия
Агрефагия, или селективная деградация белковых агрегатов посредством аутофагии, является одним из примеров перекрестного действия АЛС и УПС (рис. 5). Так, деубиквитинирующие ферменты (DUB) входят в состав обеих систем. В аутофагии также участвуют UBL-белки, которые распознаются аутофагосомными рецепторами, например SUMO-1 и FAT10 [48, 49], а также UBL-белок ISG15, который связывается с рецепторами HDAC6 и p62, содействуя лизосомному уничтожению белковых агрегатов [50]. Члены семейства молекулярных шаперонов BAG – белки BAG1 и BAG3 – конкурируют за полиубиквитинированные субстраты, связанные с шаперонами. Белок BAG1 доставляет субстраты к протеасоме, в то время как BAG3 взаимодействует непосредственно с p62 и одновременно связывает полиубиквитинированные цепи K48, направляя на деградацию в лизосомы белки, первоначально адресованные протеасоме [51]. Такие подверженные агрегации белки, как β-амилоид [52], хантингтин [53] и α-синуклеин [54], являются аутофагосомными субстратами, хотя по другим данным они также могут расщепляться протеасомой. Дрожжевой белок Cue5 является рецептором, способствующим ликвидации агрегатов, содержащих белки с полиглутаминовыми участками (polyQ). Cue5 содержит убиквитинсвязывающий домен CUE и домен AIM, опосредующий взаимодействие между убиквитинированным грузом и белками ATG8 [32]. Сверхэкспрессия белка TOLLIP человека, гомолога Cue5, который также имеет домен CUE, приводит к деградации агрегатов polyQ-белков в клеточных линиях человека [55]. У млекопитающих по меньшей мере три рецептора – SQSTM1 [28, 56], NBR1 [29] и OPTN [57] – функционируют как убиквитинсвязывающие белки, которые опосредуют взаимодействие между убиквитинированными белками и аутофагосомной машинерией. Все три рецептора имеют LIR и убиквитинсвязывающие домены, т.е. служат адаптером между белками семейства LC3/GABARAP и убиквитинированными субстратами. Предполагается, что белковые агрегаты, которые не могут подвергаться деградации УПС (например, из-за размера) могут быть уничтожены с помощью аутофагии [58].
Рис. 5. Механизм агрефагии – селективной деградации белковых субстратов посредством аутофагии
Аутолизосомный гидролиз
Закрытие аутофагосомной мембраны приводит к образованию двухслойной везикулы, называемой аутофагосомой, при созревании которой удаляются ATG-белки (рис. 6). После этого происходит слияние аутофагосомы с лизосомой, однако точный механизм процесса не до конца ясен. Известно, что в этом участвуют RAS-подобные GTP-азы, а также растворимые N-этилмалеимид-чувствительные белковые рецепторы – SNARE [15]. Кроме того, есть данные, что система микротрубочек необходима для переноса зрелых аутофагосом со случайных сайтов инициации в перинуклеарную область [59], где они сливаются с эндосомами или лизосомами. Кроме того, в регуляции переноса зрелых аутофагосом в лизосомы участвует комплекс PI3K, в котором ATG14L заменен белком, обеспечивающим устойчивость к УФ-облучению (UVRAG) [15].
После слияния с аутофагосомой кислые гидролазы в лизосоме расщепляют аутофагосомный груз, и далее питательные вещества высвобождаются обратно в цитоплазму для повторного использования клеткой. Деградация клеточного материала в лизосоме является финальной стадией аутофагии.
Рис. 6. Лизосома в аутофагосомном процессе. Лизосомы окружены однослойной мембраной, содержащей интегральные и периферические белки. Внутри лизосом расположен кислотный люмен, который содержит около 60 растворимых гидролитических ферментов и активаторов. Структурные гликопротеины LAMP1 и LAMP2 – наиболее распространенные белки лизосомной мембраны. Для поддержания устойчивой кислой среды, необходимой для внутренней гидролитической деятельности лизосомы, в лизосомной мембране активно работают вакуолярные ATP-зависимые протонные насосы V-типа (v-ATP-азы). Подобные молекулярные помпы также входят в состав LYNUS и используют энергию гидролиза ATP для перекачивания протонов в лизосомный люмен. Градиент протонов также обеспечивает перенос метаболитов, ионов и растворимых субстратов в обоих направлениях [76] и необходим для корректного транспорта вновь синтезированных лизосомных ферментов из комплекса Гольджи в лизосому
ЛИЗОСОМА
Впервые лизосомы были описаны бельгийским биохимиком Кристианом де Дюв в 1955 году [60]. Они присутствуют во всех эукариотических клетках и различаются по форме и диаметру (от 0.2 до 2.0 мкм). В настоящее время признано, что функции лизосом шире, чем считалось ранее, они участвуют во многих фундаментальных процессах, таких, как регуляция передачи сигнала и энергетический обмен, восстановление плазматической мембраны, регуляция транскрипции, клеточный гомеостаз, транспорт холестерина и иммунный ответ. Функции лизосом можно разделить на три основных типа: секреция, передача сигнала и деградация.
Лизосомы играют центральную роль в деградации клеточных органелл, внеклеточных и внутриклеточных макромолекул. Эти органеллы имеют ограниченный липидным бислоем высококислый люмен (рН ~ 4.5–5.0), который содержит пул растворимых гидролаз, способных разрушать белки, протеогликаны, нуклеиновые кислоты, а также липиды (рис. 6). Маркерным ферментом лизосом является кислая фосфатаза. Оптимум активности лизосомных ферментов приходится на pH 5.0, поэтому в нейтральной среде, например в цитоплазме, их активность сильно снижается, что защищает клетку при случайном выбросе этих ферментов из лизосомы. Тем не менее, у некоторых ферментов, особенно относящихся к классу катепсинов, внелизосомная активность сохраняется в достаточной степени, поэтому их высвобождение может влиять на клеточный метаболизм [61, 62]. Мембрана лизосом содержит белки, которые участвуют в переносе молекул как из люмена, так и в него для поддержания кислой среды, а также участвуют в слиянии лизосомы с другими клеточными структурами. Субстраты, предназначенные для деградации, попадают в лизосому различными путями. Внеклеточный материал, который необходимо подвергнуть протеолизу, доставляется в лизосому посредством эндоцитоза [63], тогда как внутриклеточные компоненты разрушаются в лизосомах благодаря аутофагии [15]. Кроме того, возможно участие лизосом в некрозе и апоптозе. Пермеабилизация лизосом и последующее высвобождение ферментов в цитозоль считаются «лизосомным апоптотическим путем». Гибель клеток, вызванная активностью лизосомных ферментов, идет по пути апоптоза или некроза в зависимости от величины пермеабилизации лизосом, а именно количества протеолитических ферментов, оказавшихся в цитозоле [64]. Так, полное разрушение органеллы с высвобождением большого количества лизосомных ферментов вызывает нерегулируемый некроз, тогда как селективная лизосомная проницаемость приводит к индукции апоптоза [65, 66]. Как только лизосомные гидролазы высвобождаются в цитозоль, они могут участвовать в апоптотическом каскаде, действуя либо совместно с каноническим каспазным путем, либо непосредственно участвуя в активном расщеплении ключевых клеточных субстратов [67, 68].
Лизосомы могут секретировать свое содержимое посредством лизосомного экзоцитоза, процесса, который можно обнаружить по перемещению маркеров лизосомной мембраны, например LAMP1 (lysosome-associated membrane protein), в плазматическую мембрану [69–71]. Этот процесс наиболее активен в некоторых типах клеток, например, в клетках гемопоэтической линии, остеокластах и меланоцитах. Лизосомы сливаются с плазматической мембраной при помощи механизма, при котором происходит активация лизосомного Ca2+-зависимого канала MCOLN1, что приводит к высвобождению содержимого лизосом во внеклеточное пространство [71–73]. Этот процесс играет важную роль в восстановлении секреции и плазматической мембраны. Первоначально считалось, что лизосомный экзоцитоз встречается только в профессиональных секреторных клетках, которые содержат лизосомоподобные органеллы (lysosome-related organelles, LRO) [74], но вскоре было показано, что клетка любого типа может выполнять данную функцию [71]. Лизосомный экзоцитоз опосредует несколько физиологических процессов, таких, как дегрануляция цитотоксических Т-лимфоцитов [75], резорбция кости остеокластами [76], защита от паразитов тучными клетками и эозинофилами [77, 78], а также функцию меланоцитов при пигментации [79] и тромбоцитов в коагуляции [80].
В молекулярный механизм, опосредующий Ca2+-регулируемый экзоцитоз лизосом, вовлечены белок семейства SNARE VAMP7, трансмембранный Ca2+-связывающий белок синаптотагмин VII (SYTVII), SNAP23, синтаксин 4 [81], а также несколько белков RAB на поверхности лизосомы [70, 82, 83]. Белки аутофагии также могут регулировать лизосомный экзоцитоз. Например, липидация аутофагосомного маркера LC3 необходима для секреции содержимого лизосомы во внеклеточное пространство, так как это приводит к направлению лизосомы на слияние с плазматической мембраной [84, 85]. Тем не менее, аутофагосомы не могут опосредовать этот процесс [85]. Интересно, что лизосомный экзоцитоз контролируется транскрипционным фактором EB (TFEB, Transcription factor EB) – главным регулятором биогенеза лизосом. TFEB способствует как стыковке, так и слиянию лизосом с плазматической мембраной, регулируя экспрессию определенных генов, белковые продукты которых способствуют опосредуемому белком MCOLN1 увеличению количества внутриклеточных ионов Ca2+ [86].
Лизосомный экзоцитоз не только отвечает за секрецию лизосомного содержимого, он также играет решающую роль в восстановлении плазматической мембраны. Повреждения плазматической мембраны приводят к быстрой миграции лизосом к поврежденным сайтам. Затем лизосомы сливаются с плазматической мембраной и эффективно запечатывают поврежденные участки [87, 88]. Этот процесс особенно важен в защитных механизмах против бактериальных инфекций [89].
Не так давно стало очевидно, что лизосома играет важную роль в определении питательных веществ и в сигнальных путях, которые участвуют в обмене веществ и росте клеток. Примечательно, что мультимолекулярный сигнальный комплекс mTORC1, главный контролер роста клеток и организмов [90], активируется на лизосомной поверхности факторами роста или в ответ на накопление аминокислот [91]. mTOR, основной каталитический компонент mTORC1, представляет собой атипичную серин/треониновую киназу, его функции часто нарушаются при различных заболеваниях и, в частности, при злокачественных образованиях [92]. Показано, что свободные аминокислоты инициируют транслокацию комплекса mTORC1 в лизосомы, где он активируется благодаря взаимодействию с Rag GTP-азой, а также белками Ragulator и Rheb, прикрепленными к лизосомной мембране [91]. Активированный mTORC1 отвечает за фосфорилирование и последующее накопление ядерного фактора TFEB в цитозоле, тем самым передавая сигналы от лизосом к ядру [93].
Строение лизосомы
Лизосомы окружены однослойной мембраной, содержащей интегральные и периферические белки. Внутри лизосом расположен кислотный люмен, который содержит около 60 растворимых гидролитических ферментов и активаторов [94], таких, как сульфатазы, гликозидазы, пептидазы, фосфатазы, липазы и нуклеазы, которые позволяют лизосоме разрушать обширный репертуар биологических субстратов, включая гликозаминогликаны, сфинголипиды, гликоген и белки [95]. Внутренний периметр лизосомной мембраны выстилает толстый гликокаликсный слой, защищающий мембрану от кислых гидролаз люмена. Растворимые ферменты люмена участвуют непосредственно в деградации, мембрана лизосом активно участвует в поддержании целостности плазматической мембраны клетки, кислотности лизосомного люмена (рН), а также в переносе метаболитов, ионов и растворимых субстратов в лизосомы и из них. Структурные гликопротеины LAMP1 и LAMP2 – наиболее распространенные белки лизосомной мембраны, они составляют более 50% от общего количества белка в этой мембране, их экспрессия варьирует в разных тканях, что говорит о различиях функций. Эти белки, особенно LAMP2, являются важными регуляторами созревания фагосом и аутофагосом, а недостаток этих белков приводит к нарушению опосредованного динеином переноса лизосом в перинуклеарное пространство, где происходит их слияние с аутофагосомами [96, 97].
Кроме того, изоформа LAMP2A участвует в аутофагии, опосредованной шаперонами (chaperone-mediated autophagy, CMA), – процессе, при котором специфические белки направляются на деградацию лизосомами посредством распознавания определенного мотива в их аминокислотной последовательности [98].
Белковый состав лизосом
В механизме распознавания лизосомой питательных веществ (lysosomal nutrient sensing, LYNUS) участвует большое число белковых комплексов, расположенных на поверхности лизосомы (рис. 6). Их роль заключается в непосредственном определении содержания питательных веществ (в частности, аминокислот) в лизосомном люмене, а также в передаче информации в цитоплазму и ядро. Для поддержания устойчивой кислой среды, необходимой для внутренней гидролитической деятельности лизосомы, в лизосомной мембране активно работают вакуолярные ATP-зависимые протонные насосы V-типа (v-ATP-азы). Подобные молекулярные помпы также входят в состав LYNUS и используют энергию гидролиза ATP для перекачивания протонов в лизосомный люмен. Градиент протонов также обеспечивает перенос метаболитов, ионов и растворимых субстратов в обоих направлениях [99], он необходим для корректного транспорта вновь синтезированных лизосомных ферментов из комплекса Гольджи в лизосому. В поддержание кислой среды лизосомного люмена также вероятно вовлечены белки семейства CLC (chloride channel), а именно CLC7 [100, 101], а также катионный канал муколипин 1 (MCOLN1, известный как TRPML1), и каналы TPC (Two-Pore Channel – двухпоровые каналы) [100], которые опосредуют перенос ионов Ca2+ и Na+ из лизосомы. Недавно идентифицированный лизосомный мембранный белок LAAT1 участвует в транспорте аминокислот лизина и аргинина из лизосомы и в нее. Этот белок, по-видимому, играет решающую роль в гомеостазе аминокислот в клетке [102, 103]. Расположенный на лизосомной мембране эндолизосомный ATP-чувствительный Na+-проницаемый канал, lysoNaATP, также участвует в распознавании питательных веществ, регуляции стабильности pH лизосомного люмена и гомеостаза аминокислот, реагируя на уровень ATP и контролируя потенциал лизосомной мембраны [104]. Необходимо отметить, что роль каждого из этих каналов и точные механизмы, которые лежат в основе регуляции подкисления лизосомного люмена, еще недостаточно изучены. Известно, что диссипация трансмембранного протонного градиента приводит к снижению эффективности транспорта через лизосомную мембрану, что, в свою очередь, ведет к ухудшению деградации клеточных отходов и, в конечном итоге, к метаболическим расстройствам [96].
Известно несколько попыток проанализировать белковый состав лизосом [94, 105]. Однако методы выделения лизосом из клетки основаны либо на субклеточном фракционировании, либо на специфических особенностях растворимых лизосомных белков, таких, как модификация маннозо-6-фосфата (Man-6-P), поэтому при анализе данных затруднительно отличить резидентные белки лизосомы от белков, направленных в лизосому на деградацию. На сегодняшний день известно около 100 лизосомных белков, 70 из которых белки лизосомного люмена и около 50 – белки лизосомной мембраны [94]. Очевидно, что, вероятнее всего, идентифицированы далеко не все лизосомные белки.
Образование лизосом
Первичные лизосомы образуются в области аппарата Гольджи, а лизосомные белки в свою очередь синтезируются и подвергаются гликозилированию в шероховатом эндоплазматическом ретикулуме (шЭПР). Стадия созревания лизосомных белков является специфической. В двухстадийной реакции концевые остатки маннозы (Man) фосфорилируются по положению C6, что происходит в цис-области Гольджи. Сначала N-ацетилглюкозамин-1-фосфат переносится на OH-группу у атома C6 в концевом остатке маннозы, затем N-ацетилглюкозамин отщепляется, а белок приобретает концевую маннозо-6-фосфатную группу [106]. Именно эта модификация обуславливает направленный транспорт лизосомных ферментов к лизосомам, а также их способность к секреции. В мембранах транс-сети Гольджи (trans-Golgi network, TGN) содержатся рецепторные молекулы для связывания Man-6-P (мannose 6-phosphate receptor, MPR). Рецепторные молекулы, распознающие Man-6-P, бывают двух типов: катионнезависимый MPR (CI-MPR) и катионзависимый MPR (CD-MPR). Они распознают лизосомные белки, несущие эти группы, и связывают их. Локальное накопление этих рецепторов происходит при участии клатрина, поэтому только специфические участки мембраны отделяются и переносятся с помощью транспортных везикул к эндолизосомам, из которых в результате созревания и образуются первичные лизосомы. В конечном итоге от остатка Man-6-P отщепляется фосфатная группа. Известным признаком созревания эндосомы до лизосомы считается постепенное закисление (до рН ~ 5) в зрелой лизосоме – именно низкое значение pH в эндолизосомах способствует отделению рецепторов Man-6-P от связанных с ними белков; затем при помощи транспортных везикул рецепторы переносятся обратно в комплекс Гольджи [95] либо подвергаются гидролизу в лизосомном люмене.
До сих пор отсутствуют исчерпывающие данные о структурной и функциональной организации лизосом, а также о механизмах, которые обеспечивают их взаимодействие с другими клеточными компартментами. Более того, не вполне ясно, как меняется состав и функциональность лизосом на протяжении жизни клетки, а также в разных тканях и органах. Кроме того, пул лизосом, судя по некоторым данным, является гетерогенным: эти органеллы, по-видимому, обладают разными механизмами поддержания внутренней кислой среды и получения метаболических сигналов [107, 108]. Эти различия, вероятно, могут быть связаны с различным позиционированием лизосом в клетках, что контролируется специализированными белковыми комплексами на поверхности лизосом, а также активностью ионных каналов [109, 110].
Роль лизосом в патологиях человека
Многие заболевания связаны со снижением активности лизосом и, следовательно, с накоплением внутриклеточного материала (например, липофусцина и убиквитина), нарушение активности лизосом наблюдается при возрастных изменениях [111]. С дефектами лизосомных ферментов связан целый ряд наследственных заболеваний, известных как лизосомные болезни накопления (lysosomal storage diseases, LSD). Описано более 50 различных типов LSD, вызванных мутациями в генах, кодирующих лизосомные растворимые гидролазы, мембранные белки или вспомогательные белки лизосом, что приводит к блокированию отдельного лизосомного катаболического пути [112]. Предполагается, что накопление одного основного субстрата обусловлено дефицитом определенного лизосомного фермента. В настоящее время эта концепция наиболее популярна, однако, большое количество данных, полученных на моделях заболеваний, а также с помощью клинических исследований, свидетельствует, что патология LSD более сложна, чем первоначально предполагалось. Клинические проявления данных заболеваний неоднородны – как системные, так и неврологические признаки могут возникать в разном возрасте и прогрессировать с разной скоростью. Чаще всего нарушаются процессы расщепления гликогена (гликогенозы), липидов (липидозы) и протеогликанов (мукополисахаридозы). Нерасщепленные макромолекулы или продукты расщепления накапливаются в лизосомах и со временем вызывают необратимое повреждение клеток. Увеличивается размер органов, что в тяжелых случаях приводит к нарушению их функции. Типичными примерами таких заболеваний являются болезнь Гоше, связанная с нарушением расщепления глюкоцереброзидов, синдром Тея–Сакса (нарушение расщепления ганглиозидов) и болезнь Помпе (нарушение расщепления гликогена). Способность MPR распознавать лизосомные ферменты, модифицированные Man-6-P, рассматривается как основа для ферментной заместительной терапии LSD [113]. Недостаток или мутации в мембранных лизосомных белках также являются факторами развития множества заболеваний. Так, недостаточное количество белка MCOLN1 вызывает муколипидоз типа IV [114]. CIC7 связан с развитием остеопетроза [115]. Мутации в белке LAMP2A вызывают болезнь Данона, при которой происходит накопление аутофагических вакуолей в мышечных клетках [116]. Белок лизосомной мембраны NPC1 (Niemann-Pick C1) участвует в экспорте холестерина из лизосомы, мутации в этом белке считаются причиной болезни Ниманна–Пика типа С [117].
Также существуют множественные свидетельства, что дисфункция лизосомы является одним из основных механизмов, лежащих в основе патогенеза нейродегенеративных заболеваний, таких, как болезни Паркинсона, Альцгеймера и Хантингтона [118, 119]. Кроме того, нежелательные для клетки белковые агрегаты могут влиять на эффективность аутофагии путем ингибирования распознавания груза, направленного на деградацию аутофагосомы [120, 121].
ЗАКЛЮЧЕНИЕ
На настоящий момент очевидно, что, помимо участия в деградации, АЛС принимает непосредственное участие во многих важных клеточных процессах, таких, как определение наличия питательных веществ, передача сигнала и регуляции метаболизма клетки. Несмотря на более чем полувековую историю изучения данной органеллы, до сих пор остаются не ясными вопросы, связанные с ее строением и работой. Cистематические подходы, такие, как транскриптомика, протеомика и метаболомика, в сочетании с биохимическими методами могут помочь выявить все компоненты лизосомы и расширить понимание функционирования АЛС в целом [122]. К сожалению, мало известно и о том, как изменяются лизосомные функции в разных клетках, тканях, на определенных стадиях клеточного развития и у разных организмов, а также в изменяющихся физиологических условиях. Кроме того, открытыми остаются вопросы существования лизосом со специализированными функциями, а также роли АЛС в патогенезе заболеваний человека, таких, как нарушение метаболизма липидов, инфекции и старение. Вдумчивое и глубокое изучение функций АЛС определенно выведет человечество на качественно новый уровень борьбы со множеством социально значимых заболеваний.
Работа выполнена при поддержке гранта РНФ № 14-14-00585-П «Молекулярный механизм убиквитин-независимого протеолиза белков протеасомой и его роль в норме и патологии».
Об авторах
Анна Кудряева
Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН
Автор, ответственный за переписку.
Email: anna.kudriaeva@gmail.com
Россия, Москва
А. Соколов
Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН
Email: anna.kudriaeva@gmail.com
Россия, Москва
Алексей Белогуров
Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН; Московский государственный университет им. М.В. Ломоносова
Email: anna.kudriaeva@gmail.com
Россия, Москва
Список литературы
- Lilienbaum A. // Int. J. Biochem. Mol. Biol. 2013. V. 4. № 1. P. 1–26.
- Hershko A., Ciechanover A., Varshavsky A. // Nat. Med. 2000. V. 6. № 10. P. 1073–1081.
- Evnouchidou I., van Endert P. // Hum. Immunol. 2019. V. 80. № 5. P. 290–295.
- Erales J., Coffino P. // Biochim. Biophys. Acta. 2014. V. 1843. № 1. P. 216–221.
- Murakami Y., Matsufuji S., Kameji T. // Nature. 1992. V. 360. № 6404. P. 597–599.
- Belogurov A., Kudriaeva A., Kuzina E., Smirnov I., Bobik T., Ponomarenko N., Kravtsova-Ivantsiv Y., Ciechanover A., Gabibov A. // J. Biol. Chem. 2014. V. 289. № 25. P. 17758–17766.
- Kudriaeva A., Kuzina E.S., Zubenko O., Smirnov I. V., Belogurov A. // FASEB J. 2019. P. fj.201802237R.
- Pierce N.W., Kleiger G., Shan S., Deshaies R.J. // Nature. 2009. V. 462. № 7273. P. 615–619.
- Kuma A., Mizushima N. // Semin. Cell Dev. Biol. 2010. V. 21. № 7. P. 683–690.
- Kriegenburg F., Ungermann C., Reggiori F. // Curr. Biol. 2018. V. 28. № 8. P. R512–R518.
- Lamb C.A., Yoshimori T., Tooze S.A. // Nat. Rev. Mol. Cell Biol. 2013. V. 14. № 12. P. 759–774.
- Grumati P., Dikic I. // J. Biol. Chem. 2018. V. 293. № 15. P. 5404–5413.
- Mizushima N. // Nat. Cell Biol. 2018. V. 20. № 5. P. 521–527.
- Wen X., Klionsky D.J. // J. Mol. Biol. 2016. V. 428. № 9. P. 1681–1699.
- Bento C.F., Renna M., Ghislat G., Puri C., Ashkenazi A., Vicinanza M., Menzies F.M., Rubinsztein D.C. // Annu. Rev. Biochem. 2016. V. 85. № 1. P. 685–713.
- Oku M., Sakai Y. // BioEssays. 2018. V. 40. № 6. P. 1800008.
- Kaushik S., Cuervo A.M. // Trends Cell Biol. 2012. V. 22. № 8. P. 407–417.
- Towers C.G., Thorburn A. // EBioMedicine. 2016. V. 14. P. 15–23.
- Son S.M., Park S.J., Lee H., Siddiqi F., Lee J.E., Menzies F.M., Rubinsztein D.C. // Cell Metab. 2019. V. 29. № 1. P. 192–201.e7.
- Rogov V., Dötsch V., Johansen T., Kirkin V. // Mol. Cell. 2014. V. 53. № 2. P. 167–178.
- Kirkin V., McEwan D.G., Novak I., Dikic I. // Mol. Cell. 2009. V. 34. № 3. P. 259–269.
- Kamber R.A., Shoemaker C.J., Denic V. // Mol. Cell. 2015. V. 59. № 3. P. 372–381.
- Lazarou M., Sliter D.A., Kane L.A., Sarraf S.A., Wang C., Burman J.L., Sideris D.P., Fogel A.I., Youle R.J. // Nature. 2015. V. 524. № 7565. P. 309–314.
- Stolz A., Ernst A., Dikic I. // Nat. Cell Biol. 2014. V. 16. № 6. P. 495–501.
- Khaminets A., Behl C., Dikic I. // Trends Cell Biol. 2016. V. 26. № 1. P. 6–16.
- Stolz A., Dikic I. // Mol. Cell. 2014. V. 56. № 3. P. 341–342.
- Husnjak K., Dikic I. // Annu. Rev. Biochem. 2012. V. 81. P. 291–322.
- Pankiv S., Clausen T.H., Lamark T., Brech A., Bruun J.-A., Outzen H., Øvervatn A., Bjørkøy G., Johansen T. // J. Biol. Chem. 2007. V. 282. № 33. P. 24131–24145.
- Kirkin V., Lamark T., Sou Y.-S., Bjørkøy G., Nunn J.L., Bruun J.-A., Shvets E., McEwan D.G., Clausen T.H., Wild P., et al. // Mol. Cell. 2009. V. 33. № 4. P. 505–516.
- Korac J., Schaeffer V., Kovacevic I., Clement A.M., Jungblut B., Behl C., Terzic J., Dikic I. // J. Cell Sci. 2013. V. 126. № 2. P. 580–592.
- Zhou J., Wang J., Cheng Y., Chi Y.-J., Fan B., Yu J.-Q., Chen Z. // PLoS Genet. 2013. V. 9. № 1. P. e1003196.
- Lu K., Psakhye I., Jentsch S. // Cell. 2014. V. 158. № 3. P. 549–563.
- Zheng Y.T., Shahnazari S., Brech A., Lamark T., Johansen T., Brumell J.H. // J. Immunol. 2009. V. 183. № 9. P. 5909–5916.
- Thurston T.L.M., Ryzhakov G., Bloor S., von Muhlinen N., Randow F. // Nat. Immunol. 2009. V. 10. № 11. P. 1215–1221.
- Wild P., Farhan H., McEwan D.G., Wagner S., Rogov V.V., Brady N.R., Richter B., Korac J., Waidmann O., Choudhary C., et al. // Science. 2011. V. 333. № 6039. P. 228–233.
- Deosaran E., Larsen K.B., Hua R., Sargent G., Wang Y., Kim S., Lamark T., Jauregui M., Law K., Lippincott-Schwartz J., et al. // J. Cell Sci. 2013. V. 126. № 4. P. 939–952.
- Sarraf S.A., Raman M., Guarani-Pereira V., Sowa M.E., Huttlin E.L., Gygi S.P., Harper J.W. // Nature. 2013. V. 496. № 7445. P. 372–376.
- Wong Y.C., Holzbaur E.L.F. // Proc. Natl. Acad. Sci. USA. 2014. V. 111. № 42. P. E4439–E4448.
- Grasso D., Ropolo A., Lo Ré A., Boggio V., Molejón M.I., Iovanna J.L., Gonzalez C.D., Urrutia R., Vaccaro M.I. // J. Biol. Chem. 2011. V. 286. № 10. P. 8308–8324.
- Marshall R.S., Li F., Gemperline D.C., Book A.J., Vierstra R.D. // Mol. Cell. 2015. V. 58. № 6. P. 1053–1066.
- Pohl C., Jentsch S. // Nat. Cell Biol. 2009. V. 11. № 1. P. 65–70.
- Guo H., Chitiprolu M., Gagnon D., Meng L., Perez-Iratxeta C., Lagace D., Gibbings D. // Nat. Commun. 2014. V. 5. № 1. P. 5276.
- Watson R.O., Manzanillo P.S., Cox J.S. // Cell. 2012. V. 150. № 4. P. 803–815.
- Morimoto D., Walinda E., Fukada H., Sou Y.-S., Kageyama S., Hoshino M., Fujii T., Tsuchiya H., Saeki Y., Arita K., et al. // Nat. Commun. 2015. V. 6. № 1. P. 6116.
- Linares J.F., Duran A., Yajima T., Pasparakis M., Moscat J., Diaz-Meco M.T. // Mol. Cell. 2013. V. 51. № 3. P. 283–296.
- Olzmann J.A., Li L., Chudaev M.V., Chen J., Perez F.A., Palmiter R.D., Chin L.-S. // J. Cell Biol. 2007. V. 178. № 6. P. 1025–1038.
- Collins G.A., Goldberg A.L. // Cell. 2017. V. 169. № 5. P. 792–806.
- Kalveram B., Schmidtke G., Groettrup M. // J. Cell Sci. 2008. V. 121. № 24. P. 4079–4088.
- Cho S.-J., Yun S.-M., Jo C., Lee D., Choi K.J., Song J.C., Park S.I., Kim Y.-J., Koh Y.H. // Autophagy. 2015. V. 11. № 1. P. 100–112.
- Nakashima H., Nguyen T., Goins W.F., Chiocca E.A. // J. Biol. Chem. 2015. V. 290. № 3. P. 1485–1495.
- Gamerdinger M., Kaya A.M., Wolfrum U., Clement A.M., Behl C. // EMBO Rep. 2011. V. 12. № 2. P. 149–156.
- Pickford F., Masliah E., Britschgi M., Lucin K., Narasimhan R., Jaeger P.A., Small S., Spencer B., Rockenstein E., Levine B., et al. // J. Clin. Invest. 2008. V. 118. № 6. P. 2190–2199.
- Ravikumar B., Vacher C., Berger Z., Davies J.E., Luo S., Oroz L.G., Scaravilli F., Easton D.F., Duden R., O’Kane C.J., et al. // Nat. Genet. 2004. V. 36. № 6. P. 585–595.
- Winslow A.R., Chen C.-W., Corrochano S., Acevedo-Arozena A., Gordon D.E., Peden A.A., Lichtenberg M., Menzies F.M., Ravikumar B., Imarisio S., et al. // J. Cell Biol. 2010. V. 190. № 6. P. 1023–1037.
- Lu K., Psakhye I., Jentsch S. // Autophagy. 2015. V. 10. № 12. P. 2381–2382.
- Ohsumi Y., Ichimura Y., Kirisako T., Takao T., Satomi Y., Shimonishi Y., Ishihara N., Mizushima N., Tanida I., Kominami E., et al. // Nature. 2000. V. 408. № 6811. P. 488–492.
- Shen Z., Li Y., Gasparski A.N., Abeliovich H., Greenberg M.L. // J. Biol. Chem. 2017. V. 292. № 7. P. 2916–2923.
- Korolchuk V.I., Menzies F.M., Rubinsztein D.C. // FEBS Lett. 2010. V. 584. № 7. P. 1393–1398.
- Jahreiss L., Menzies F.M., Rubinsztein D.C. // Traffic. 2008. V. 9. № 4. P. 574–587.
- de Duve C. // Nat. Cell Biol. 2005. V. 7. № 9. P. 847–849.
- Saftig P., Schröder B., Blanz J. // Biochem. Soc. Trans. 2010. V. 38. № 6. P. 1420–1423.
- Olson O.C., Joyce J.A. // Nat. Rev. Cancer. 2015. V. 15. № 12. P. 712–729.
- Luzio J.P., Parkinson M.D.J., Gray S.R., Bright N.A. // Biochem. Soc. Trans. 2009. V. 37. Pt 5. P. 1019–1021.
- Li W., Yuan X., Nordgren G., Dalen H., Dubowchik G.M., Firestone R.A., Brunk U.T. // FEBS Lett. 2000. V. 470. № 1. P. 35–39.
- Bursch W. // Cell Death Differ. 2001. V. 8. № 6. P. 569–581.
- Guicciardi M.E., Leist M., Gores G.J. // Oncogene. 2004. V. 23. № 16. P. 2881–2890.
- Leist M., Jäättelä M. // Cell Death Differ. 2001. V. 8. № 4. P. 324–326.
- Leist M., Jäättelä M. // Nat. Rev. Mol. Cell Biol. 2001. V. 2. № 8. P. 589–598.
- Chieregatti E., Meldolesi J. // Nat. Rev. Mol. Cell Biol. 2005. V. 6. № 2. P. 181–187.
- Verhage M., Toonen R.F. // Curr. Opin. Cell Biol. 2007. V. 19. № 4. P. 402–408.
- Rodríguez A., Webster P., Ortego J., Andrews N.W. // J. Cell Biol. 1997. V. 137. № 1. P. 93–104.
- Andrews N.W. // Trends Cell Biol. 2000. V. 10. № 8. P. 316–321.
- Jaiswal J.K., Andrews N.W., Simon S.M. // J. Cell Biol. 2002. V. 159. № 4. P. 625–635.
- Luzio J.P., Hackmann Y., Dieckmann N.M.G., Griffiths G.M. // Cold Spring Harb. Perspect. Biol. 2014. V. 6. № 9. P. a016840.
- Stinchcombe J.C., Griffiths G.M. // Annu. Rev. Cell Dev. Biol. 2007. V. 23. № 1. P. 495–517.
- Mostov K., Werb Z. // Science. 1997. V. 276. № 5310. P. 219–220.
- Logan M.R., Odemuyiwa S.O., Moqbel R. // J. Allergy Clin. Immunol. 2003. V. 111. № 5. P. 923–932; quiz 933.
- Wesolowski J., Paumet F. // Immunol. Res. 2011. V. 51. № 2–3. P. 215–226.
- Stinchcombe J., Bossi G., Griffiths G.M. // Science. 2004. V. 305. № 5680. P. 55–59.
- Ren Q., Ye S., Whiteheart S.W. // Curr. Opin. Hematol. 2008. V. 15. № 5. P. 537–541.
- Rao S.K., Huynh C., Proux-Gillardeaux V., Galli T., Andrews N.W. // J. Biol. Chem. 2004. V. 279. № 19. P. 20471–20479.
- Jahn R., Scheller R.H. // Nat. Rev. Mol. Cell Biol. 2006. V. 7. № 9. P. 631–643.
- Bossi G., Griffiths G. // Semin. Immunol. 2005. V. 17. № 1. P. 87–94.
- Cadwell K., Liu J.Y., Brown S.L., Miyoshi H., Loh J., Lennerz J.K., Kishi C., Kc W., Carrero J.A., Hunt S., et al. // Nature. 2008. V. 456. № 7219. P. 259–263.
- DeSelm C.J., Miller B.C., Zou W., Beatty W.L., van Meel E., Takahata Y., Klumperman J., Tooze S.A., Teitelbaum S.L., Virgin H.W. // Dev. Cell. 2011. V. 21. № 5. P. 966–974.
- Medina D.L., Fraldi A., Bouche V., Annunziata F., Mansueto G., Spampanato C., Puri C., Pignata A., Martina J.A., Sardiello M., et al. // Dev. Cell. 2011. V. 21. № 3. P. 421–430.
- Gerasimenko J.V., Gerasimenko O.V., Petersen O.H. // Curr. Biol. 2001. V. 11. № 23. P. R971–R974.
- Reddy A., Caler E.V., Andrews N.W. // Cell. 2001. V. 106. № 2. P. 157–169.
- Roy D., Liston D.R., Idone V.J., Di A., Nelson D.J., Pujol C., Bliska J.B., Chakrabarti S., Andrews N.W. // Science. 2004. V. 304. № 5676. P. 1515–1518.
- Laplante M., Sabatini D.M. // Cell. 2012. V. 149. № 2. P. 274–293.
- Sancak Y., Bar-Peled L., Zoncu R., Markhard A.L., Nada S., Sabatini D.M. // Cell. 2010. V. 141. № 2. P. 290–303.
- Guertin D.A., Sabatini D.M. // Cancer Cell. 2007. V. 12. № 1. P. 9–22.
- Settembre C., Zoncu R., Medina D.L., Vetrini F., Erdin S.S., Erdin S.S., Huynh T., Ferron M., Karsenty G., Vellard M.C., et al. // EMBO J. 2012. V. 31. № 5. P. 1095–1108.
- Schröder B.A., Wrocklage C., Hasilik A., Saftig P. // Proteomics. 2010. V. 10. № 22. P. 4053–4076.
- Luzio J.P., Pryor P.R., Bright N.A. // Nat. Rev. Mol. Cell Biol. 2007. V. 8. № 8. P. 622–632.
- Saftig P., Klumperman J. // Nat. Rev. Mol. Cell Biol. 2009. V. 10. № 9. P. 623–635.
- Saftig P., Beertsen W., Eskelinen E.-L. // Autophagy. 2008. V. 4. № 4. P. 510–512.
- Alessandrini F., Pezzè L., Ciribilli Y. // Semin. Oncol. 2017. V. 44. № 4. P. 239–253.
- Marshansky V., Rubinstein J.L., Grüber G. // Biochim. Biophys. Acta - Bioenerg. 2014. V. 1837. № 6. P. 857–879.
- Mindell J.A. // Annu. Rev. Physiol. 2012. V. 74. № 1. P. 69–86.
- Chakraborty K., Leung K., Krishnan Y. // Elife. 2017. V. 6. e28862
- Liu B., Du H., Rutkowski R., Gartner A., Wang X. // Science. 2012. V. 337. № 6092. P. 351–354.
- Efeyan A., Zoncu R., Sabatini D.M. // Trends Mol. Med. 2012. V. 18. № 9. P. 524–533.
- Cang C., Zhou Y., Navarro B., Seo Y., Aranda K., Shi L., Battaglia-Hsu S., Nissim I., Clapham D.E., Ren D. // Cell. 2013. V. 152. № 4. P. 778–790.
- Bagshaw R.D., Mahuran D.J., Callahan J.W. // Mol. Cell. Proteomics. 2005. V. 4. № 2. P. 133–143.
- Coutinho M.F., Prata M.J., Alves S. // Mol. Genet. Metab. 2012. V. 105. № 4. P. 542–550.
- Korolchuk V.I., Saiki S., Lichtenberg M., Siddiqi F.H., Roberts E.A., Imarisio S., Jahreiss L., Sarkar S., Futter M., Menzies F.M., et al. // Nat. Cell Biol. 2011. V. 13. № 4. P. 453–460.
- Johnson D.E., Ostrowski P., Jaumouillé V., Grinstein S. // J. Cell Biol. 2016. V. 212. № 6. P. 677–692.
- Pu J., Schindler C., Jia R., Jarnik M., Backlund P., Bonifacino J.S. // Dev. Cell. 2015. V. 33. № 2. P. 176–188.
- Li X., Rydzewski N., Hider A., Zhang X., Yang J., Wang W., Gao Q., Cheng X., Xu H. // Nat. Cell Biol. 2016. V. 18. № 4. P. 404–417.
- Rubinsztein D.C., Mariño G., Kroemer G. // Cell. 2011. V. 146. № 5. P. 682–695.
- Futerman A.H., van Meer G. // Nat. Rev. Mol. Cell Biol. 2004. V. 5. № 7. P. 554–565.
- Neufeld E.F. // Birth Defects Orig. Artic. Ser. 1980. V. 16. № 1. P. 77–84.
- Bach G., Bargal R., Avidan N., Ben-Asher E., Olender Z., Zeigler M., Frumkin A., Raas-Rothschild A., Glusman G., Lancet D. // Nat. Genet. 2000. V. 26. № 1. P. 118–123.
- Sobacchi C., Schulz A., Coxon F.P., Villa A., Helfrich M.H. // Nat. Rev. Endocrinol. 2013. V. 9. № 9. P. 522–536.
- Nascimbeni A.C., Fanin M., Angelini C., Sandri M. // Cell Death Dis. 2017. V. 8. № 1. P. e2565–e2565.
- Lloyd-Evans E., Morgan A.J., He X., Smith D.A., Elliot-Smith E., Sillence D.J., Churchill G.C., Schuchman E.H., Galione A., Platt F.M. // Nat. Med. 2008. V. 14. № 11. P. 1247–1255.
- Menzies F.M., Fleming A., Rubinsztein D.C. // Nat. Rev. Neurosci. 2015. V. 16. № 6. P. 345–357.
- Guo F., Liu X., Cai H., Le W. // Brain Pathol. 2018. V. 28. № 1. P. 3–13.
- Orenstein S.J., Kuo S.-H., Tasset I., Arias E., Koga H., Fernandez-Carasa I., Cortes E., Honig L.S., Dauer W., Consiglio A., et al. // Nat. Neurosci. 2013. V. 16. № 4. P. 394–406.
- Martinez-Vicente M., Talloczy Z., Wong E., Tang G., Koga H., Kaushik S., de Vries R., Arias E., Harris S., Sulzer D., et al. // Nat. Neurosci. 2010. V. 13. № 5. P. 567–576.
- Walkley S.U. // J. Inherit. Metab. Dis. 2009. V. 32. № 2. P. 181–189.
Дополнительные файлы